Publicado: 27/05/25 16:08 Categorías: Biología molecular
NGS 2.0. Nuevas tecnologías, nuevas fronteras
La secuenciación de nueva generación (NGS) ya no es tan “nueva”, puesto que ha pasado a ser una herramienta clave en investigación básica y un aliado incuestionable ampliamente utilizado en diversas áreas; diagnóstico, terapias personalizadas, vigilancia ambiental o alimentaria, biotecnología de cultivos o salud animal, entre otras.
Si bien los cimientos de esta técnica ya están bien asentados y la tecnología es cada vez más accesible, en los últimos años este campo está siendo testigo de un crecimiento vertiginoso y una expansión en las capacidades iniciales de secuenciación. Muestras que antes quedaban fuera del rango analizable mediante NGS ahora tienen soluciones técnicas sofisticadas con una sensibilidad, resolución y escalabilidad sin precedentes.
.png)
La técnica ha sido ampliamente utilizada en diferentes ámbitos como en el análisis de restos de fosas comunes de la guerra civil, fósiles de especies perdidas y muestras parafinadas de hace décadas, así como, en la secuenciación de células individuales y embriones de 3 días de antigüedad.
La especificidad del trabajo de muestras con NGS ha avanzado, de conocer la información genética de una célula, hasta conocer su ubicación dentro de un tejido, a través del uso de las ómicas espaciales.
Ahora somos capaces de secuenciar más profundamente y mejor con nuevas alternativas tecnológicas y de equipamiento. Pasamos de limitarnos a secuenciaciones de fragmentos cortos, a trabajar con la obtención de lecturas de varios kb, que facilitan el ensamblaje de novo del genoma de especies no estudiadas, o el análisis de isoformas de genes de interés.
La necesidad de procesar numerosas muestras en paralelo también ha dado lugar al auge de las soluciones de automatización, para minimizar el error humano y permitir el procesamiento rápido y preciso de cientos de muestras a la vez. Además, la NGS ya no sólo se realiza en laboratorios especializados con equipamiento altamente complejo y costoso, sino que existen opciones al alcance de cualquier laboratorio y que puedan usarse incluso in situ.
Evolución de las tecnologías NGS
Aunque durante años el mercado estuvo monopolizado por un par de marcas, en la actualidad contamos ya con numerosas tecnologías alternativas y/o complementarias:
Lecturas largas (long-read sequencing): las plataformas de Oxford Nanopore (MinION, GridION, PromethION) o PacBio (SMRT/HiFi), cada vez más extendidas, permiten lecturas de ADN de miles a decenas de múltiples bases, hasta 100 veces más largas que las obtenidas por tecnologías tradicionales de lectura corta.
Esto mejora enormemente el ensamblaje del genoma y la detección de reordenamientos complejos. Por ejemplo, en algunos tipos de cáncer la secuenciación de lectura larga ha sido clave para entender variantes de fusión génica (como BCR-ABL1) que influyen en la respuesta al tratamiento.
Nuevos jugadores: BGI/MGI ha desarrollado sistemas de secuenciación basados en DNBseq, una tecnología que usa círculos de ADN para lograr alta precisión y rendimiento. Su modelo MGISEQ-T7 de alto rendimiento puede procesar cuatro flow cells simultáneas, generando hasta 6 TB de datos por día (equivalente a unos 60 genomas humanos completos diarios).
Estas soluciones compiten en velocidad y coste con Illumina, ofreciendo una alternativa emergente muy atractiva en el mercado.
Secuenciación de muestras complejas: la tecnología de Takara Bio, ThruPLEX DNA-Seq, permite obtener librerías secuenciables e indexadas de alta complejidad en un solo tubo en 2 horas, desde apenas 50 pg de ADN.
De forma similar, los kits SMARTer (Clontech/Takara) permiten generar librerías de RNA-Seq a partir de picogramos de ARN, o directamente desde célula única, sin necesidad de purificación previa del ARN.
Estas soluciones han hecho viable la secuenciación de muestras muy preciadas y delicadas (biopsias líquidas, ARN degradado, ADN inmunoprecipitado) en entornos clínicos y forenses.
Ómicas espaciales: la irrupción de tecnologías y equipos de célula única como 10X Chromium o BD Rhapsody han supuesto un avance sin precedentes en el área de la genómica. Ahora, además de conocer el transcriptoma de las neuronas de un corte cerebral puedo saber dónde se está expresando cada gen en ese tejido.
Tecnologías actuales, como Curio Trekker de Takara Bio, combinan NGS con información espacial tisular y ofrecen la posibilidad de marcar en 1 hora núcleos individuales con etiquetas espaciales, para luego poder convertir los datos genómicos (UMAP) en un mapa espacial del tejido de origen con una resolución de 10 micras.
Estas tecnologías permiten obtener información epigenética, genómica o transcriptómica de alta calidad, a nivel unicelular, respetando la arquitectura tisular y abriendo nuevas fronteras en la caracterización de tumores, la neurociencia o la biología del desarrollo.
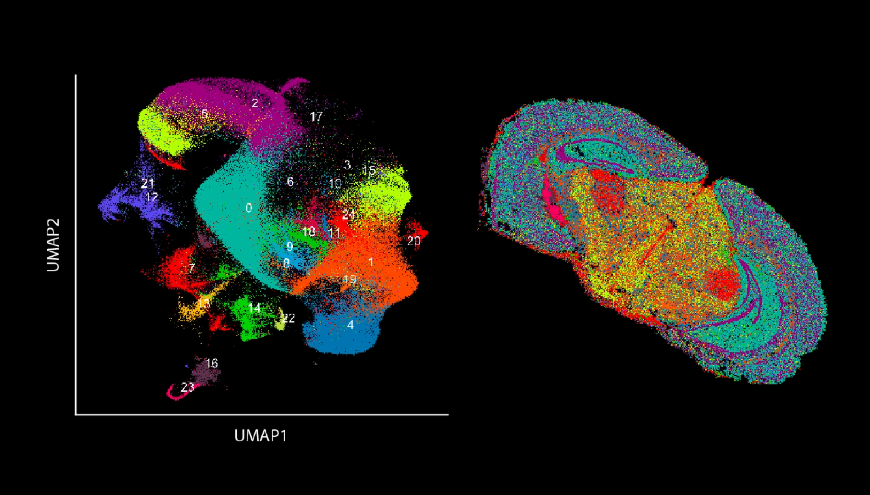
Figura: Curio Bioscience permite transformar información ómica de célula única en un mapa de expresión con resolución de 10 micras.
Nuevas perspectivas para la NGS
El futuro de la secuenciación masiva apunta a una mayor velocidad, accesibilidad y aplicaciones integradas. La inteligencia artificial es clave para potenciar la integración de datos multi-ómicos (genómica con transcriptómica, epigenómica, proteómica, etc.), y facilitar la aplicación clínica de tecnologías espaciales y de célula única y la interpretación de los crecientes volúmenes de datos.
La convergencia de secuenciación masiva, análisis bioinformático avanzado y biología de vanguardia augura un futuro donde la información genética será un componente central y transversal en la salud, la investigación, la agricultura y la conservación ambiental.
Condalab, en su compromiso con el avance tecnológico en secuenciación genómica, continúa desarrollando su catálogo de soluciones NGS e incorpora nuevos productos y equipamiento para dar respuesta a los principales retos del sector.
Descarga nuestro catálogo actualizado y descubre nuevas soluciones para estudios de ómica espacial, reactivos para secuenciación por MGI y ONT y automatización de librerías NGS.

 Probióticos: ¿Cuál es el bueno?
Probióticos: ¿Cuál es el bueno?
 Virus recombinantes en investigación y bioproducción: retos y soluciones
Virus recombinantes en investigación y bioproducción: retos y soluciones
 Condalab dice SÍ a la feria líder en tecnologías del laboratorio: Analytica 2026
Condalab dice SÍ a la feria líder en tecnologías del laboratorio: Analytica 2026
 CONDALAB estará presente en WHX Labs Dubai 2026
CONDALAB estará presente en WHX Labs Dubai 2026
 Condalab y BD: una colaboración que impulsa las ómicas espaciales
Condalab y BD: una colaboración que impulsa las ómicas espaciales