Publicado: 14/10/20 10:33 Categorías: Biología molecular
Desde Condalab, hemos hablado en numerosas ocasiones de este sistema revolucionario; sin embargo, sus aplicaciones no dejan de sorprendernos día tras día.
La última de ellas se ha producido en el campo de la biología del desarrollo, y viene de la mano de científicos del Centro Andaluz de Biología del Desarrollo (CABD), en colaboración con el Stowers Institute de Estados Unidos, que han empleado la tecnología CRISPR/Cas para estudiar el papel del RNA materno en las primeras horas del desarrollo embrionario.
La mayoría de los estudios llevados a cabo con CRISPR se basan en la capacidad de estas tijeras moleculares para cortar el DNA; sin embargo, se ha identificado una nueva nucleasa, la Cas13 que, a diferencia de la Cas9, es capaz de cortar RNA.
La primera versión de esta proteína fue descrita por Feng Zhang en el año 2017, quien la aisló de la bacteria Leptotrichia wadei (LwaCas13a). Esta nucleasa presenta actividad RNAsa, que se activa gracias a la presencia de una molécula de RNA guía y que, una vez activada, digiere el RNA presente.
Miguel A. Moreno, investigador del CABD, aprovechó las características de este sistema para poder implementarlo en diversos modelos animales, de tal modo que pudiese emplearse no solo para cortar el RNA, sino también para modificarlo o para ayudar a localizarlo en el interior celular.
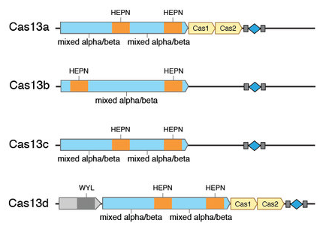
Pero para poder desarrollar la herramienta óptima, el Dr. Moreno tuvo que probar diversas nucleasas del tipo Cas13, procedentes de distintas bacterias, pues no todas funcionaban igual en cultivos celulares y en modelos animales. Así, de todas las que probaron, solo una presentaba parámetros de eficiencia y de especificidad compatibles con lo que ellos buscaban, la RfxCas13d, derivada de la especie Ruminococcus flavefaciens.
El empleo de esta proteína les permitió obtener un sistema útil para la depleción específica de transcritos de mRNA en embriones animales, herramienta de gran utilidad en el estudio de la embriogénesis, concretamente en el proceso conocido como transición de madre a cigoto, o MZT.
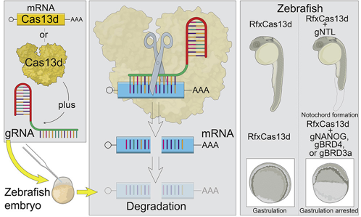
Mediante este proceso, los mRNA maternos se degradan, siendo reemplazados por elementos del genoma del cigoto, por lo que su estudio resulta de vital importancia para conocer exactamente cuáles de los factores maternos depositados en el ovocito tienen un papel fundamental en los primeros pasos del desarrollo del cigoto.
Los resultados han sido publicados en la prestigiosa revista Development Cell, en un artículo titulado “CRISPR-Cas13 Induces Efficient mRNA Knockdown in Animal Embryos”.
De hecho, este estudio ha servido de base para un proyecto financiado por el Consejo Superior de Investigaciones Científicas, cuyo objetivo es la aplicación del sistema CRISPR-Cas para la destrucción del genoma del SARS-CoV-2, el coronavirus causante de la COVID-19.
Si quieres conocer todo lo que podemos ofrecerte desde Condalab para trabajar con la herramienta CRISPR/Cas, puedes consultar nuestro workflow para experimentos de knock-out y knock-in o echar un vistazo a nuestra webinar al respecto. También puedes informarte sobre los métodos diagnósticos que se están desarrollando para detectar la COVID-19 empleando el sistema CRISPR en este enlace.
Bibliografía:

 Probióticos: ¿Cuál es el bueno?
Probióticos: ¿Cuál es el bueno?
 Virus recombinantes en investigación y bioproducción: retos y soluciones
Virus recombinantes en investigación y bioproducción: retos y soluciones
 Condalab dice SÍ a la feria líder en tecnologías del laboratorio: Analytica 2026
Condalab dice SÍ a la feria líder en tecnologías del laboratorio: Analytica 2026
 CONDALAB estará presente en WHX Labs Dubai 2026
CONDALAB estará presente en WHX Labs Dubai 2026
 Condalab y BD: una colaboración que impulsa las ómicas espaciales
Condalab y BD: una colaboración que impulsa las ómicas espaciales