Publicado: 21/05/20 09:14 Categorías: Biología molecular
Las aplicaciones de CRISPR en el diagnóstico del SARS-CoV-2
Durante los últimos meses, todas las miradas están puestas en un mismo foco: el SARS-CoV-2. Son muchos los esfuerzos que se están llevando a cabo para luchar contra este virus, tanto a nivel diagnóstico (buscando nuevas formas de detección precoz) como de tratamiento.
Las técnicas más conocidas para diagnosticar el COVID-19 son, por un lado, la Reacción en Cadena de la Polimerasa (PCR) y, por otra, la prueba serológica o de detección de anticuerpos.
Mientras que la PCR permite localizar y amplificar un fragmento del ARN viral dando, por tanto, un resultado cuantitativo sobre los niveles de infección, los denominados coloquialmente como “test rápidos” detectan anticuerpos generados frente a dicho virus (o, en algunos casos, antígenos específicos del mismo). Estos últimos sistemas son mucho más rápidos que la PCR pero, por contra, presentan una sensibilidad mucho menor y no permiten el diagnóstico precoz, al ser necesaria una ventana de tiempo para que el sistema inmunitario del paciente genere los anticuerpos frente al virus.
Pero, ¿y si pudiésemos juntar las ventajas de ambas técnicas, la sensibilidad y la rapidez, para obtener un método de diagnóstico precoz? Parece una utopía, pero ya son varios los grupos que trabajan en su desarrollo empleando como base la tecnología CRISPR/Cas.
CRISPR/Cas es una herramienta molecular que permite la edición del genoma de cualquier célula aprovechando, para ello, la capacidad de ciertas proteínas para cortar el ADN. Pero, ¿cómo trasladar este método al diagnóstico del nuevo coronavirus?
Ya en 2017, el laboratorio estadounidense dirigido por el Dr. Feng Zhang desarrolló un método de diagnóstico mediante CRISPR, al que denominó SHERLOCK (Specific High-sensitivity Enzymatic Reporter un-LOCKing). Dicho método se basa en el empleo de una nucleasa distinta a la Cas9 y que, a diferencia de esta, tiene capacidad para cortar el ARN, la proteína Cas13a.
Al combinar esta proteína con un ARN guía (diseñado contra la secuencia específica del gen a degradar), los científicos vieron que la RNasa degradaba no sólo las secuencias target, como se esperaba, sino todo los ARN que encontraba a su paso. Lejos de tomarlo como un fracaso, Zhang desarrolló el método SHERLOCK con el fin de detectar la presencia de moléculas de ARN (y ADN) en una muestra fuera cual fuera su concentración en la misma. ¿Cómo? Pues simplemente añadiendo unas moléculas de ARN que en sus extremos presentan una molécula fluorescente y otra inhibidora de la fluorescencia, muy similares a las empleadas en el diseño de sondas de tipo Taqman.
Cuando estas dos moléculas están juntas, la fluorescencia está inhibida pero, ante la presencia del fragmento diana, la Cas13a ejerce su efecto y corta no solo los ARN de la muestra, sino también los añadidos exógenamente posibilitando, de este modo, que las moléculas se separen y se desinhiba la fluorescencia. Pero este corte sólo se producirá si la nucleasa es capaz de ejercer su acción y, para ello, necesita que el ARN guía se una a su secuencia diana. De este modo, sólo se podrá detectar la fluorescencia si el fragmento de interés se encuentra en la muestra analizada; de lo contrario, la Cas13a no podrá cortar y la fluorescencia seguirá inhibida.
La gran ventaja de este método reside en la especificidad y sensibilidad que presenta, ya que permite detectar cantidades del orden de attomolar (hasta 10-18 molar). Es por ello que, ante la creciente amenaza del SARS-CoV-2, este grupo de científicos elaboró y publicó un protocolo de detección de dicho genoma viral mediante la metodología SHERLOCK.
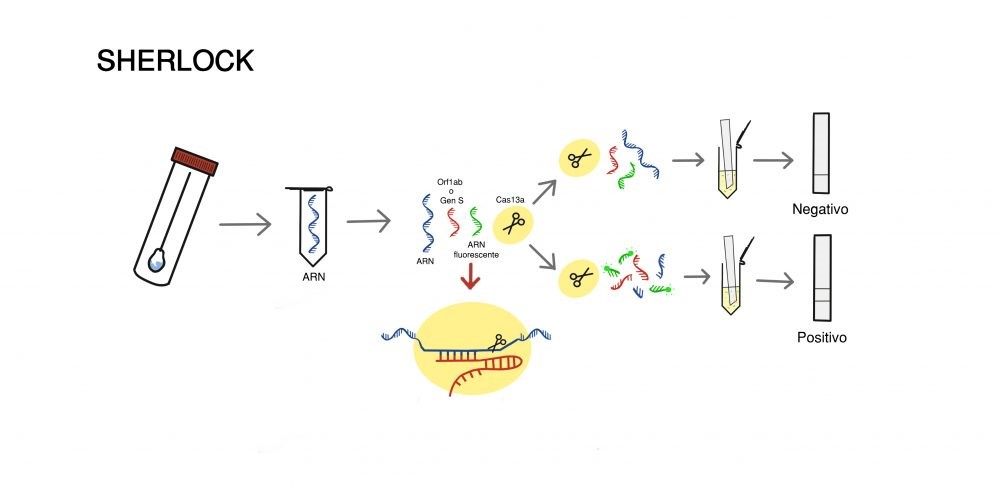
Dicho protocolo consta de 3 etapas, y puede completarse en apenas 1 hora.
- Amplificación isotérmica del ARN extraído de la muestra (25 minutos a 42oC).
- Detección del ARN viral mediante la incubación del mismo con la nucleasa Cas13, el RNA guía y el RNA reporter (30 minutos a 37oC).
- Lectura visual de los resultados (en tira, 5 minutos)
La prueba está diseñada de tal manera que permita la detección de los genes de la proteína S (o spike protein, implicada en la entrada del virus) y del gen Orf1ab (que codifica una replicasa del virus).
No obstante, se trataba de un protocolo meramente experimental hasta que, a principios de mayo, dada la situación de alarma global, la empresa Sherlock Biosciences (fundada, entre otros, por el Dr. Zhang) recibió la autorización de la FDA para uso en emergencias para su kit SherlockTM CRISPR SARS-CoV-2, que se encuentra actualmente en desarrollo.
De forma casi paralela a dicha aprobación, este mismo grupo hizo público un nuevo método de diagnóstico basado en la misma tecnología pero de forma simplificada. Bajo el nombre de STOPCovid, esta técnica permite detectar el SARS-CoV-2 de forma directa, sin necesidad de realizar la extracción o aislamiento del ARN y tras una única incubación a 60oC. Con un formato similar al de los test serológicos, los resultados ofrecen una elevada sensibilidad y especificad en apenas una hora; sin embargo, a diferencia de la metodología anterior, ésta aún no está aprobada para su uso en clínica, por lo que su aplicación se limita al ámbito de la investigación.
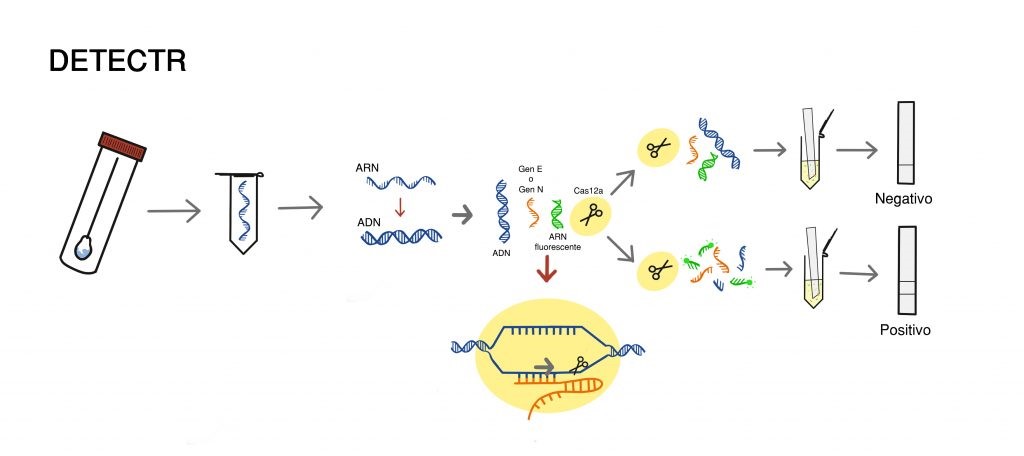
Pero SHERLOCK no es el único método existente para el diagnóstico mediante CRISPR. Otros laboratorios, como el de Jennifer Doudna (pionera en el descubrimiento de CRISPR), también han trabajado y puesto a punto un protocolo basado en esta misma herramienta, al que han bautizado como DETECTR (DNA Endonuclease TargEted CRISPR Trans Reporter). Al igual que el anterior, este también puede ser aplicado en la detección del nuevo coronavirus aunque su uso sigue estando, por ahora, meramente destinado a la investigación.
Son muchos los esfuerzos que se están llevando a cabo actualmente para luchar contra esta pandemia. Desde Condalab, queremos colaborar en todo lo posible y, por ello, ponemos a tu disposición un amplio abanico de productos para la investigación y detección del SARS-CoV-2. También ofrecemos múltiples herramientas relacionadas con la metodología CRISPR/Cas.
Para más información, no dudes en ponerte en contacto con nosotros a través del siguiente enlace, estaremos encantados de ayudarte.
Fuente:

 Probióticos: ¿Cuál es el bueno?
Probióticos: ¿Cuál es el bueno?
 Virus recombinantes en investigación y bioproducción: retos y soluciones
Virus recombinantes en investigación y bioproducción: retos y soluciones
 Condalab dice SÍ a la feria líder en tecnologías del laboratorio: Analytica 2026
Condalab dice SÍ a la feria líder en tecnologías del laboratorio: Analytica 2026
 CONDALAB estará presente en WHX Labs Dubai 2026
CONDALAB estará presente en WHX Labs Dubai 2026
 Condalab y BD: una colaboración que impulsa las ómicas espaciales
Condalab y BD: una colaboración que impulsa las ómicas espaciales